Quien haya tenido que trabajar con librerías de Inteligencia Artificial y Deep Learning, como lo son TensorFlow y Keras, sabrá que tarde o temprano va a necesitar sacarle el máximo provecho a su equipo si quiere acelerar las cosas.
Entrenar tus modelos con un la CPU, por potente que esta sea, puede llevar incluso días, y por supuesto no queremos eso. Por suerte, hasta en un portátil normalito como el mio, es posible acelerar mucho las cosas (y dejarlo en unas horas) si entrenamos esos mismos modelos usando la capacidad de cómputo de nuestra GPU.
Conseguirlo puede no ser una tarea fácil, y es que aunque estas librerías de Artificial Intelligence vienen perfectamente preparadas para ello, pelearse con entornos de Python, pip, Conda, NVIDIA, CUDA, cuDNN y Windows puede convertirse en una auténtica pesadilla. Tenemos la opción de contenerizarlo todo y tener entornos limpios, aislados y versionados, gracias a Docker, que además se ve muy potenciado si utilizamos WSL (Subsistema de Windows para Linux) en nuestra máquina Windows.
Si necesitas aprovechar la potencia de tu GPU para TensorFlow y Keras en tus cuadernos de Jypter, en un entorno Windows, aquí tienes los pasos que seguí yo, con la esperanza de que te sirvan de ayuda.
Paso a paso
Requisitos
- Windows 10 o posterior
- Hyper-V activo en tu BIOS
- Una tarjeta gráfica NVIDIA compatible con CUDA (en teoria todas las posteriores al 2008 lo serían).
Paso 1. Instalación de CUDA en Windows
En primer lugar necesitamos instalar los drivers adecuados para WSL y nuestra versión de Windows, que son los que nos permiten enviar procesos a la GPU para aprovechar su potencia de cómputo.
Puedes descargarlos en este enlace a CUDA WSL, yo instalé la versión Studio. Después instalalos normalmente siguiendo las instrucciones del propio instalador.
Paso 2. Instalación de WSL2 (Subsistema de Windows para Linux)
En PowerShell, ejecutar:
wsl –install
Si ya lo tenias instalado, o si quieres asegurarte, puedes probar a actualizarlo:
wsl –update
Establece la versión 2 como predeterminada para evitar problemas despues con las IPTABLES en Docker.
wsl –set-default-version 2
En este punto te recomiendo reiniciar tu máquina, aunque arriesgarte a seguir sin hacerlo como hice yo jaja.
Paso 3. Instalación de Ubuntu en Windows con WSL
Vamos a instalar una distribución de Linux en Windows usando WSL2, en concreto yo instalé Ubuntu-20.04. De nuevo en PowerShell:
wsl –install -d Ubuntu-20.04
Asegurate de que tu distribución se ha instalado con la versión 2 de WSL, lo puedes comprobar con el siguiente comando:
wsl -l -v
Paso 4. Iniciar sesión en Ubuntu
El siguiente comando abrirá una consola conectada a nuestro nuevo entorno Linux.
wsl -d Ubuntu-20.04
La primera vez necesitarás crear un usuario y contraseña. ¡Trata de no olvidarlos!
Paso 5. Instalación de dependencias en Linux
En nuestra sesión de Linux (que acabamos de iniciar), vamos a instalar las dependencias que necesitamos. En primer lugar, Instalaremos Docker:
curl https://get.docker.com sh
Una vez haya terminado, instalaremos también dentro de Linux, el NVIDIA Container toolkit:
distribution=$(. /etc/os-release;echo $ID$VERSION_ID)
curl -s -L https://nvidia.github.io/nvidia-docker/gpgkey | sudo apt-key add -
curl -s -L https://nvidia.github.io/nvidia-docker/$distribution/nvidia-docker.list | sudo tee /etc/apt/sources.list.d/nvidia-docker.list
sudo apt-get update
sudo apt-get install -y nvidia-docker2
Paso 6. Comprobar el servicio de Docker
Con el siguiente comando podemos comprobar que docker está correctamente instalado y en ejecución:
sudo service docker status
Si está en ejecución, perfecto, puedes pasar al Paso 7. si no fuese así, puede deberse a varias razones:
Posibilidad 2: El servicio no se ha iniciado automaticamente
Parece ser que en WSL a veces el servicio no arranca automaticamente, puedes probar reiniciando el demonio manualmente
sudo dockerd
O, como hice yo, iniciar una nueva ventana en la misma instalación de linux lanzando en PowerShell de nuevo el comando “wsl -d Ubuntu-20.04”, y parando y relanzando el servicio desde ahí:
sudo service docker stop
sudo service docker start
Posibilidad 2: Reiniciar
En serio, prueba a reiniciar y luego vuelve a ejecutar el service docker start anterior. A mi siempre me ha bastado con esto.
Posibilidad 3: El servicio no se inicia por un problema con las IPTABLES y la red
Mira el Paso 3 y usa el comando que aparece ahí para asegurarte de que estás usando la versión 2 de WSL.
wsl -l -v
Posibilidad 4: Alguna otra razón
Pueden pasar muchas cosas, como que se esté intentando ocupar una IP que en tu red esté ya en uso y eso impida que el servicio se inicie. WSL tiene algunos problemas en la forma en que maneja la red, y eso da bastante guerra al usar docker. En este punto lo unico que puedes es comprobar el log de docker para tirar del hilo:
sudo cat /var/log/docker.log
Mucho ánimo, ya casi lo tienes!
Paso 7. Comprobar la instalación
Si todo está en orden, podemos descargar y ejecutar un contenedor de docker que utilice las GPUs disponibles para hacer un benchmark de rendimiento.
sudo docker run –gpus all nvcr.io/nvidia/k8s/cuda-sample:nbody nbody -gpu -benchmark
Si todo funciona correctamente, habremos conseguido ejecutar un contenedor de docker dentro de linux dentro de windows, que accede a nuestra GPU!. ¿Maravilloso no? El resultado debería ser similar a este, donde se muestra nuestro dispositivo y la velocidad alcanzada:
> Compute 6.1 CUDA device: [NVIDIA GeForce GTX 1050 Ti]
6144 bodies, total time for 10 iterations: 5.173 ms
= 72.969 billion interactions per second
= 1459.382 single-precision GFLOP/s at 20 flops per interaction
También probé con otro portatil con una gráfica más potente y aquí puede verse la diferencia.
> Compute 7.5 CUDA device: [NVIDIA GeForce GTX 1650]
16384 bodies, total time for 10 iterations: 25.335 ms
= 105.955 billion interactions per second
= 2119.095 single-precision GFLOP/s at 20 flops per interaction
Si escribimos el comando:
nvidia-smi
Podemos obtener un resumen con la versión del Driver, la versión de CUDA, y los procesos en ejecución en la GPU.
+-----------------------------------------------------------------------------+
| NVIDIA-SMI 515.75 Driver Version: 517.40 CUDA Version: 11.7 |
|-------------------------------+----------------------+----------------------+
| GPU Name Persistence-M| Bus-Id Disp.A | Volatile Uncorr. ECC |
| Fan Temp Perf Pwr:Usage/Cap| Memory-Usage | GPU-Util Compute M. |
| | | MIG M. |
|===============================+======================+======================|
| 0 NVIDIA GeForce ... On | 00000000:01:00.0 Off | N/A |
| N/A 43C P0 N/A / N/A | 0MiB / 4096MiB | 1% Default |
| | | N/A |
+-------------------------------+----------------------+----------------------+
+-----------------------------------------------------------------------------+
| Processes: |
| GPU GI CI PID Type Process name GPU Memory |
| ID ID Usage |
|=============================================================================|
| No running processes found |
+-----------------------------------------------------------------------------+
Paso 8. Lanzar el Notebook de Jupyter desde el contenedor de Docker
Al igual que hemos hecho con el benchmark, también podemos descargar y ejecutar un contenedor de docker con TensorFlow y Jupyter preinstalados.
sudo docker run -it –gpus all -p 8888:8888 tensorflow/tensorflow:latest-gpu-py3-jupyter
Al final de la ejecución, la traza nos mostrará un enlace a localhost que si abrimos en un navegador de nuestra máquina, nos permitirá acceder a nuestro Jupyter Notebook.
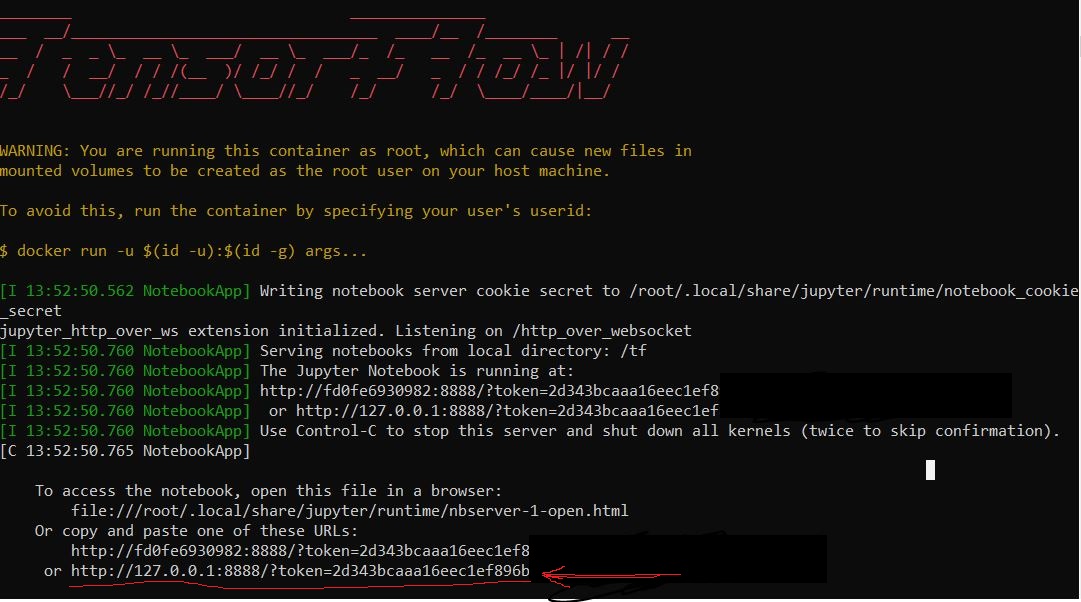
Por defecto Jupyter se ejecuta en el puerto 8888, pero si ya tuviesemos ese puerto ocupado en nuestra máquina, podemos mapearlo en otro cambiando el 8888:8888 por un nuevo puerto, por ejemplo 9080:8888.
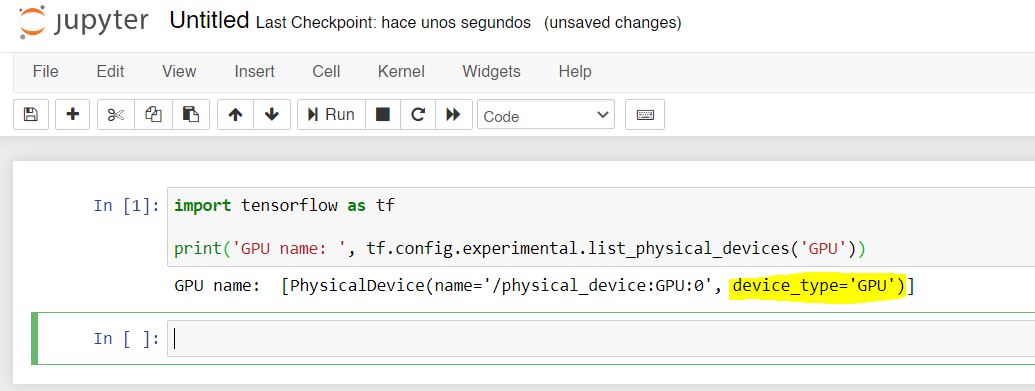
Ahí podemos ejecutar el siguiente código de Python para comprobar que TensorFlow puede acceder a la GPU:
import tensorflow as tf
print('GPU name: ', tf.config.experimental.list_physical_devices('GPU'))
Paso 9 (Opcional). Compartir ficheros de nuestra máquina con Jupyter.
La máquina host (Windows) comparte los ficheros con WSL (Linux) mediante una unidad de red. La forma más facil de abrir esta carpeta compartida para mover ahí algunos ficheros y tenerlos disponibles en Linux es escribiendo el siguiente comando en nuestra consola Linux.
explorer.exe .
Esto abrira un explorador de ficheros (No olvides el punto al final del comando que indica que quieres abrir la carpeta actual). Ahí puedes copiar los ficheros que quieras tener accesibles en Linux. Suponiendo que no hayas cambiado de ruta, seguirás en el directorio de ${HOME}. Puedes conocer tu carpeta de trabajo actual escribiendo:
pwd
Ahora puedes crear un volumen en docker que mapea tu carpeta de Linux con la carpeta de trabajo de Jupyter dentro del contenedor de Docker (/tf/notebooks). Por ejemplo, imagina que en tu ${HOME} has creado una subcarpeta “notebooks” y en ella has puesto tus cuadernos de Jypter. Pues podrías mapearlo así:
sudo docker run -it –gpus all -v ${HOME}/notebooks:/tf/notebooks -p 8888:8888 tensorflow/tensorflow:latest-gpu-py3-jupyter
Ahora, al abrir Jupyter en el navegador, encontrarás que puedes acceder a los cuadernos que hayas copiado.
Nota: es posible que falle si ya tenias el puerto ocupado, por ejemplo porque hayas ejecutado otro contenedor antes y no lo has detenido antes de lanzar este. No me voy a enredar con como manejar docker porque eso daría para otro artículo, pero siempre puedes recurrir al Ctrl+C para interrumpirlo :). Aquí te dejo una referencia rápida de comandos de Docker.
Paso 10 (Opcional). Crear un contenedor Docker con nuestras propias dependencias y paquetes de Python.
Si además de TensorFlow necesitas otras librerías o paquetes, como Scikit-learn, Numpy, Pandas, o Keras, puedes instalar los paquetes en el contenedor que acabamos de descargar, o lo que yo recomendaría: crear tu propia imagen de Docker con las dependencias que necesites. Así podrás tener un contendor limpio y preparado para trabajar siempre que te haga falta.
Para ello, abre de nuevo la carpeta de Linux con
explorer.exe .
Y crea en ella un documento de definición de imagen de Docker o Dockerfile. El nombre debe ser “Dockerfile” sin extensión. Editalo a tu antojo para que incluya las dependencias que necesites. Dejo aquí el mio de ejemplo.
# Imagen base
FROM tensorflow/tensorflow:2.9.2-gpu-jupyter
## instalar tus propias dependencias
RUN apt-get update
RUN apt install graphviz -y
RUN pip install scikit-learn
RUN pip install torch
RUN pip install theano
RUN pip install pandas
RUN pip install keras
RUN pip install numpy
RUN pip install matplotlib
RUN pip install scipy
RUN pip install summary
Una vez guardado el fichero, vuelve a Linux y, en la ruta donde está el fichero Dockerfile, compila la imagen y dale un nombre:
docker build -t minombre/miimagen:mitag .
Finalmente, puedes ejecutar tu flamante contenedor personalizado con un comando similar al que veniamos usando, pero empleando tu imagen en su lugar:
sudo docker run -it –gpus all -v ${HOME}/notebooks:/tf/notebooks -p 8888:8888 minombre/miimagen:mitag
Si quieres más información, incluso acerca de como publicar tu nueva imagen en Dockerhub para compartirla, este artículo puede serte de gran ayuda.
Conclusión
Espero que este tutorial paso a paso te sirva de ayuda. A mi me llevo mucho tiempo en su primer momento para conseguir una instalación que funcionase, ya que pensaba que podría utilizar CUDA desde Windows con Docker instalado de manera nativa, pero al parecer no era así. Si al final de esta guía has conseguido tener un entorno funcionando, notarás una gran diferencia en el rendimiento cuando entrenes tus modelos de IA usando la GPU.
Dejo por aquí algunos enlaces y referencias que me fueron de gran ayuda durante mi propia instalación y que espero ahorraros.
Referencias principales
- Ejecución de Jupyter en Docker con la CPU.
- Muy util: Install WSL2 on Windows 11 with NVIDIA GPU and Docker Support
- Build your own docker image
- Jupyter Notebooks with NVIDIA GPU using Tensorflow and Pytorch
- Docker Stacks documentation, GPU enabled notebooks
- Docker commands cheatsheet
Otros enlaces interesantes:
- Setup any Tensorflow-gpu & Jupyter in minutes [Ubuntu 18.04]
- Instalación NVIDIA CUDA en Windows
- Instalación NVIDIA CUDNN
- Instalar WSL Ubuntu
- Fix unauthorized NVIDIA repo in docker image 2.2.3-gpu-py3-jupyter
- Instalación de TensorFlow en Docker
- How to Run Jupyter Notebook on Docker
- CUDA toolkit documentation and installation
- Using GPUs With Keras: A Tutorial With Code